
 |
| &epsilon | N |
p-value | # of modules in lean network | # of modules in heavy network |
| .35 | .222 | .020 | 1 | 1 |
| .40 | .455 | .031 | 1 | 1 |
| .45 | .793 | .019 | 3 | 1 |
| .50 | .976 | .033 | 5 | 1 |
| .55 | .999 | .001 | 3 | 1 |
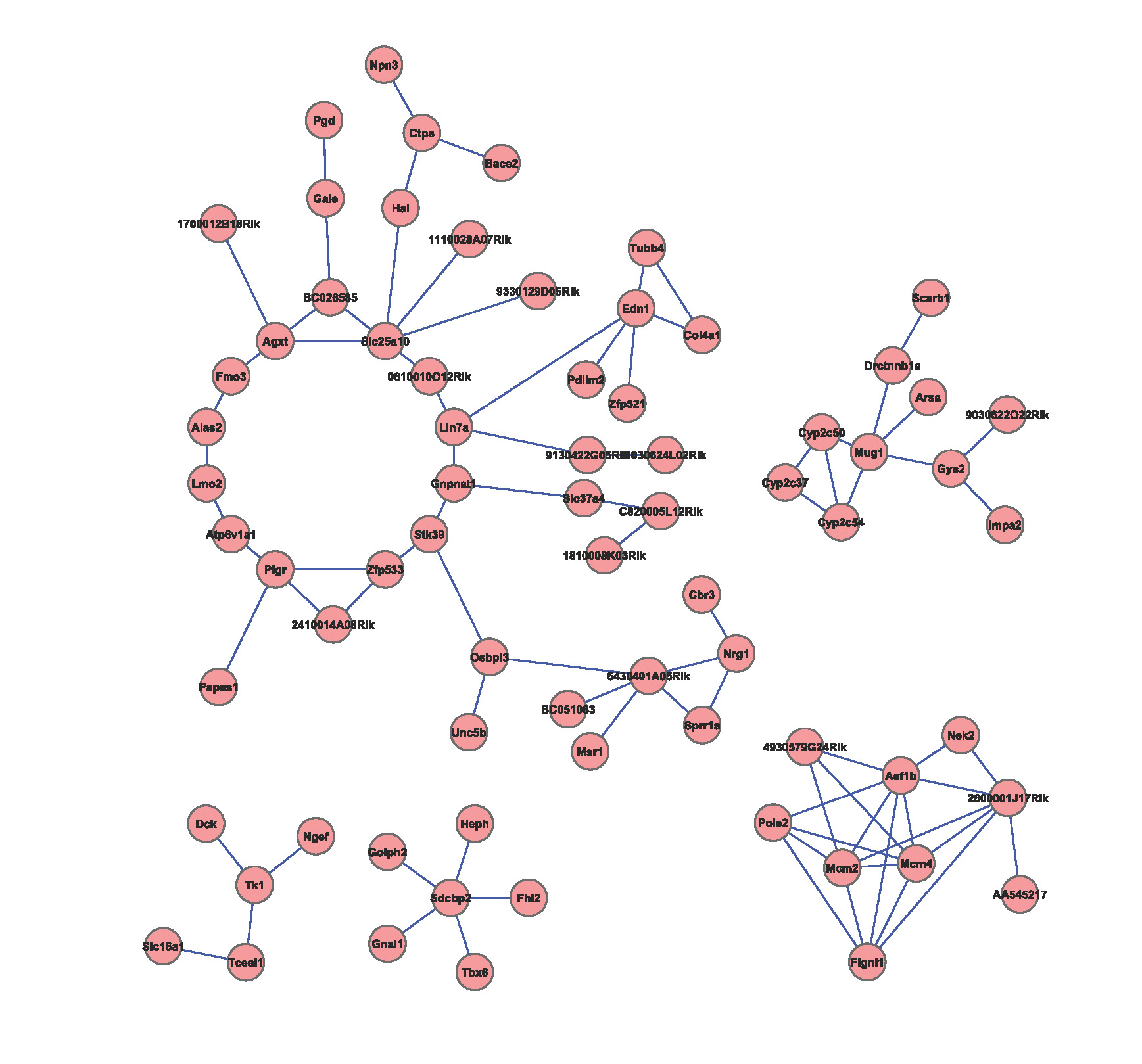

| SubstanceBXH | Gene | d | p-value |
| MMT00067823 | Anxa2 | 0.118 | 0.000 |
| MMT00056866 | Anxa5 | 0.119 | 0.000 |
| MMT00030931 | Apom | 0.186 | 0.000 |
| MMT00078851 | F7 | 0.122 | 0.000 |
| MMT00021805 | Igfbp7 | 0.157 | 0.000 |
| MMT00081331 | Itih1 | 0.149 | 0.000 |
| MMT00065159 | Kng2 | 0.168 | 0.000 |
| MMT00074736 | Scnn1a | 0.148 | 0.000 |
| MMT00055005 | Slc22a7 | 0.154 | 0.000 |
| MMT00061313 | Slc43a1 | 0.162 | 0.000 |
| MMT00048209 | Spp1 | 0.232 | 0.000 |
| MMT00078732 | 9430028I06Rik | 0.153 | 0.000 |
| MMT00056362 | AA960558 | 0.153 | 0.001 |
| MMT00050363 | Map4k4 | 0.145 | 0.001 |
| MMT00052695 | Proz | 0.126 | 0.001 |
| MMT00003081 | 2310046G15Rik | 0.158 | 0.001 |
| MMT00021649 | Erbb3 | 0.167 | 0.003 |
| MMT00006713 | Ppic | 0.097 | 0.003 |
| MMT00031263 | Tuba1 | 0.132 | 0.003 |
| MMT00026944 | Igfbp2 | 0.182 | 0.004 |
| MMT00072042 | Col14a1 | 0.161 | 0.005 |
| MMT00067261 | Serpinf2 | 0.15 | 0.005 |
| MMT00061509 | Fabp4 | 0.16 | 0.006 |
| MMT00080150 | Fkbp7 | 0.121 | 0.007 |
| MMT00055467 | Marcks | 0.139 | 0.007 |
| MMT00006300 | Tubb2 | 0.16 | 0.007 |
| MMT00074488 | Epb4.1l2 | 0.124 | 0.008 |
| MMT00024492 | F2r | 0.149 | 0.008 |
| MMT00026397 | 1200015A22Rik | 0.155 | 0.009 |
| MMT00067079 | Fetub | 0.142 | 0.010 |
| MMT00006315 | 5031439A09Rik | 0.123 | 0.011 |
| MMT00067525 | Ddah2 | 0.119 | 0.011 |
| MMT00053218 | Armcx2 | 0.155 | 0.012 |
| MMT00077345 | Maged2 | 0.146 | 0.015 |
| MMT00008463 | Pdir | 0.141 | 0.017 |
| MMT00074326 | Sdcbp | 0.134 | 0.018 |
| MMT00050107 | Trip13 | 0.193 | 0.019 |
| MMT00002209 | Tusc3 | 0.177 | 0.019 |
| MMT00064235 | Ang1 | 0.140 | 0.020 |
| MMT00056798 | 2310016C16Rik | 0.150 | 0.021 |
| MMT00006230 | Npn3 | 0.157 | 0.023 |
| MMT00032979 | Aldh1a7 | 0.137 | 0.025 |
| MMT00042972 | Aox1 | 0.165 | 0.026 |
| MMT00000840 | Col5a3 | 0.205 | 0.028 |
| MMT00062460 | Ntf3 | 0.197 | 0.030 |
| MMT00030465 | Arhgap24 | 0.169 | 0.032 |
| MMT00071189 | S100a10 | 0.151 | 0.036 |
| MMT00013203 | Mat1a | 0.149 | 0.037 |
| MMT00034198 | Cbr3 | 0.188 | 0.039 |
| MMT00018643 | C86987 | 0.188 | 0.044 |
| MMT00039457 | 2600017H08Rik | 0.161 | 0.044 |
| MMT00074541 | Nid1 | 0.138 | 0.044 |
| MMT00015563 | Ppm1l | 0.184 | 0.045 |
| MMT00036669 | Rnf11 | 0.122 | 0.047 |
| MMT00061815 | Arhgap18 | 0.156 | 0.048 |
| MMT00021004 | Xbp1 | 0.195 | 0.048 |
| MMT00075754 | 3732412D22Rik | 0.187 | 0.050 |
| MMT00061312 | Lcat | 0.166 | 0.052 |
| MMT00024307 | Tubb4 | 0.177 | 0.054 |
| MMT00064617 | Vim | 0.136 | 0.054 |
| MMT00056571 | BC026585 | 0.202 | 0.055 |
| MMT00072057 | Col4a1 | 0.165 | 0.057 |
| MMT00008968 | Ggta1 | 0.190 | 0.058 |
| MMT00055441 | Tax1bp3 | 0.129 | 0.058 |
| MMT00076621 | 2010002N04Rik | 0.190 | 0.062 |
| MMT00039459 | Fsp27 | 0.123 | 0.065 |
| MMT00031617 | Tnc | 0.192 | 0.065 |
| MMT00071976 | AI173486 | 0.140 | 0.067 |
| MMT00034467 | E130307J04Rik | 0.168 | 0.068 |
| MMT00023864 | 6720460F02Rik | 0.162 | 0.069 |
| MMT00018239 | Cyp2d13 | 0.188 | 0.071 |
| MMT00054261 | Edn1 | 0.209 | 0.072 |
| MMT00033171 | Serpina11 | 0.154 | 0.072 |
| MMT00078976 | Cd36 | 0.171 | 0.074 |
| MMT00031873 | BC024139 | 0.173 | 0.075 |
| MMT00042247 | Tpd52 | 0.157 | 0.079 |
| MMT00081439 | Ugdh | 0.136 | 0.086 |
| MMT00039465 | Clca2 | 0.182 | 0.088 |
| MMT00063772 | Adamts4 | 0.194 | 0.089 |
| MMT00060559 | Fgb | 0.132 | 0.089 |
| MMT00027064 | Gpm6b | 0.173 | 0.093 |
| MMT00056584 | Trfr2 | 0.127 | 0.094 |
| MMT00010873 | Atp8b2 | 0.101 | 0.096 |
| MMT00070040 | Dck | 0.168 | 0.097 |
| MMT00028908 | Lrp1 | 0.140 | 0.097 |
| MMT00014444 | Ap3s1 | 0.123 | 0.102 |
| MMT00054735 | BC038881 | 0.179 | 0.103 |
| MMT00025902 | Amigo | 0.158 | 0.105 |
| MMT00066141 | Mtnr1a | 0.152 | 0.106 |
| MMT00067105 | Stk39 | 0.197 | 0.106 |
| MMT00025527 | Vtn | 0.150 | 0.106 |
| MMT00053787 | Anxa3 | 0.124 | 0.118 |
| MMT00026028 | AI324046 | 0.131 | 0.120 |
| MMT00018537 | 1810023F06Rik | 0.154 | 0.121 |
| MMT00067156 | Emcn | 0.125 | 0.122 |
| MMT00037796 | 4930405M20Rik | 0.165 | 0.123 |
| MMT00007300 | Slc25a24 | 0.122 | 0.124 |
| MMT00070677 | Il1r1 | 0.164 | 0.128 |
| MMT00059089 | Cyp4f13 | 0.145 | 0.130 |
| MMT00054136 | Synpo | 0.153 | 0.132 |
| MMT00012203 | 4632428N05Rik | 0.151 | 0.134 |
| MMT00012992 | Fmo3 | 0.169 | 0.135 |
| MMT00041355 | Plk2 | 0.152 | 0.137 |
| MMT00037571 | Aldh1a7 | 0.156 | 0.142 |
| MMT00007963 | Sdcbp2 | 0.187 | 0.142 |
| MMT00044996 | Pex11a | 0.138 | 0.144 |
| MMT00059404 | 1810008K03Rik | 0.189 | 0.146 |
| MMT00072000 | Cyp2c37 | 0.137 | 0.147 |
| MMT00018607 | 1110008L20Rik | 0.161 | 0.149 |
| MMT00063359 | 1110028A07Rik | 0.148 | 0.150 |
| MMT00025633 | 3930402F23Rik | 0.134 | 0.151 |
| MMT00045090 | Gstm1 | 0.160 | 0.151 |
| MMT00058036 | 2410014A08Rik | 0.171 | 0.152 |
| MMT00025886 | Tgm1 | 0.166 | 0.154 |
| MMT00043964 | Lxn | 0.125 | 0.156 |
| MMT00027763 | Slc38a2 | 0.181 | 0.156 |
| MMT00045775 | 2600011E07Rik | 0.172 | 0.159 |
| MMT00070201 | Icam2 | 0.130 | 0.160 |
| MMT00028683 | Msx2 | 0.125 | 0.162 |
| MMT00051144 | Nid2 | 0.146 | 0.162 |
| MMT00079074 | D330012D11Rik | 0.158 | 0.163 |
| MMT00007117 | Ngef | 0.150 | 0.164 |
| MMT00065264 | Psmd8 | 0.150 | 0.165 |
| MMT00074523 | Pdlim2 | 0.206 | 0.167 |
| MMT00045980 | Scd2 | 0.123 | 0.169 |
| MMT00022959 | Abcg2 | 0.139 | 0.172 |
| MMT00063274 | Mesp1 | 0.138 | 0.174 |
| MMT00018296 | Hprt | 0.105 | 0.177 |
| MMT00082712 | Lrat | 0.142 | 0.177 |
| MMT00082461 | Thpo | 0.125 | 0.177 |
| MMT00049556 | Matn2 | 0.163 | 0.182 |
| MMT00026611 | Col1a2 | 0.145 | 0.186 |
| MMT00031229 | Avpr1a | 0.157 | 0.196 |
| MMT00025021 | Ly6d | 0.147 | 0.203 |
| MMT00062787 | Rnase4 | 0.126 | 0.205 |
| MMT00022657 | C8b | 0.129 | 0.209 |
| MMT00034709 | BC011468 | 0.134 | 0.210 |
| MMT00024538 | Raet1e | 0.188 | 0.210 |
| MMT00010083 | Cyp4f15 | 0.153 | 0.212 |
| MMT00033001 | 4930579G24Rik | 0.154 | 0.213 |
| MMT00071772 | AI428936 | 0.205 | 0.218 |
| MMT00065116 | Cdc42ep3 | 0.165 | 0.223 |
| MMT00013549 | 5830411E10Rik | 0.135 | 0.228 |
| MMT00004671 | Aqp8 | 0.116 | 0.232 |
| MMT00029192 | Lamb3 | 0.173 | 0.233 |
| MMT00035294 | BC010552 | 0.167 | 0.236 |
| MMT00081375 | Fhl2 | 0.145 | 0.242 |
| MMT00072411 | Drctnnb1a | 0.166 | 0.245 |
| MMT00007603 | Arsa | 0.161 | 0.247 |
| MMT00014558 | 9030624L02Rik | 0.148 | 0.247 |
| MMT00078969 | Slc6a12 | 0.124 | 0.248 |
| MMT00028408 | Bet1 | 0.152 | 0.249 |
| MMT00009302 | BC051083 | 0.133 | 0.250 |
| MMT00024150 | Clstn3 | 0.170 | 0.252 |
| MMT00017203 | 9-Sep | 0.180 | 0.257 |
| MMT00045707 | Sart2 | 0.158 | 0.260 |
| MMT00079130 | Alas2 | 0.194 | 0.266 |
| MMT00082869 | 1110025F24Rik | 0.175 | 0.267 |
| MMT00061484 | Dll4 | 0.130 | 0.277 |
| MMT00055415 | Slc37a4 | 0.158 | 0.278 |
| MMT00026973 | Mx1 | 0.144 | 0.280 |
| MMT00042538 | Msr1 | 0.152 | 0.281 |
| MMT00003569 | 1500004O14Rik | 0.129 | 0.286 |
| MMT00033419 | 1700093E07Rik | 0.133 | 0.287 |
| MMT00006709 | Zfp521 | 0.139 | 0.292 |
| MMT00043149 | Actg | 0.162 | 0.293 |
| MMT00049262 | 4632413K17Rik | 0.120 | 0.304 |
| MMT00074692 | Mmp13 | 0.173 | 0.305 |
| MMT00072999 | Mcm4 | 0.149 | 0.306 |
| MMT00007277 | 2600017H02Rik | 0.154 | 0.306 |
| MMT00024107 | 5730485H21Rik | 0.131 | 0.319 |
| MMT00023027 | Arid5a | 0.150 | 0.320 |
| MMT00025256 | Gmpr | 0.139 | 0.320 |
| MMT00069398 | 3110050N22Rik | 0.175 | 0.323 |
| MMT00061586 | 9430059P22Rik | 0.2 | 0.323 |
| MMT00049092 | Ccbl1 | 0.137 | 0.326 |
| MMT00078486 | Crat | 0.165 | 0.327 |
| MMT00015380 | Sprr1a | 0.118 | 0.337 |
| MMT00077659 | Gale | 0.141 | 0.340 |
| MMT00071610 | Mmp27 | 0.164 | 0.342 |
| MMT00067910 | Mlp | 0.162 | 0.344 |
| MMT00047734 | Nfkbil1 | 0.190 | 0.345 |
| MMT00059054 | Lgals1 | 0.182 | 0.346 |
| MMT00055391 | Serpina3n | 0.125 | 0.346 |
| MMT00061944 | Scarb1 | 0.143 | 0.348 |
| MMT00046920 | Mod1 | 0.115 | 0.354 |
| MMT00069588 | Phca | 0.173 | 0.355 |
| MMT00060760 | Chst7 | 0.133 | 0.358 |
| MMT00006142 | Slc16a1 | 0.160 | 0.359 |
| MMT00005542 | Spnb3 | 0.129 | 0.362 |
| MMT00061857 | Lama2 | 0.151 | 0.363 |
| MMT00063833 | Klra5 | 0.167 | 0.366 |
| MMT00046654 | Phf7 | 0.166 | 0.375 |
| MMT00015674 | Agpt2 | 0.148 | 0.377 |
| MMT00038022 | Pgd | 0.125 | 0.386 |
| MMT00028002 | Pparg | 0.175 | 0.390 |
| MMT00058021 | Dnaic1 | 0.167 | 0.391 |
| MMT00046778 | Armcx1 | 0.143 | 0.393 |
| MMT00067333 | Cml1 | 0.168 | 0.398 |
| MMT00079155 | Anxa1 | 0.168 | 0.400 |
| MMT00039035 | 6430401A05Rik | 0.169 | 0.403 |
| MMT00074983 | Gpx4 | 0.145 | 0.404 |
| MMT00073538 | 2310004K20Rik | 0.148 | 0.404 |
| MMT00039396 | Gnpnat1 | 0.170 | 0.405 |
| MMT00051321 | Kifc2 | 0.152 | 0.415 |
| MMT00080548 | Sdc4 | 0.139 | 0.416 |
| MMT00076818 | Slc25a10 | 0.176 | 0.419 |
| MMT00030014 | 9130422G05Rik | 0.149 | 0.428 |
| MMT00058679 | 1810044O22Rik | 0.128 | 0.429 |
| MMT00049893 | 9030622O22Rik | 0.166 | 0.434 |
| MMT00026438 | 2810408E11Rik | 0.142 | 0.437 |
| MMT00012202 | AW547365 | 0.183 | 0.438 |
| MMT00016084 | E430036I04Rik | 0.151 | 0.441 |
| MMT00040888 | Postn | 0.174 | 0.441 |
| MMT00067947 | Nrg1 | 0.155 | 0.442 |
| MMT00027530 | 5430432M24Rik | 0.121 | 0.443 |
| MMT00040765 | Igfals | 0.169 | 0.447 |
| MMT00010973 | Tbx6 | 0.184 | 0.466 |
| MMT00061739 | Ppm1f | 0.144 | 0.467 |
| MMT00056001 | Pole2 | 0.154 | 0.471 |
| MMT00050299 | 4931406C07Rik | 0.147 | 0.472 |
| MMT00061767 | Tceal1 | 0.129 | 0.474 |
| MMT00015524 | Hal | 0.171 | 0.476 |
| MMT00058222 | C820005L12Rik | 0.186 | 0.477 |
| MMT00007859 | Npdc1 | 0.138 | 0.489 |
| MMT00003575 | 2810439K08Rik | 0.144 | 0.496 |
| MMT00005727 | Fignl1 | 0.140 | 0.499 |
| MMT00011832 | Egr2 | 0.140 | 0.510 |
| MMT00046171 | 2510042P03Rik | 0.154 | 0.514 |
| MMT00005418 | Dp1 | 0.171 | 0.515 |
| MMT00041310 | 2310057H16Rik | 0.146 | 0.520 |
| MMT00018170 | 4933434G05Rik | 0.151 | 0.520 |
| MMT00069683 | Acat2 | 0.143 | 0.524 |
| MMT00034916 | Cyp2g1 | 0.150 | 0.536 |
| MMT00031586 | Ces2 | 0.139 | 0.539 |
| MMT00069238 | Ung | 0.156 | 0.552 |
| MMT00039138 | Lmo2 | 0.165 | 0.553 |
| MMT00063470 | Agxt | 0.181 | 0.56 |
| MMT00045746 | Spata13 | 0.150 | 0.574 |
| MMT00070540 | Sema6d | 0.154 | 0.575 |
| MMT00017837 | Impa2 | 0.172 | 0.582 |
| MMT00048978 | Cyp2c50 | 0.145 | 0.593 |
| MMT00041568 | Zfp533 | 0.158 | 0.599 |
| MMT00026166 | AA545217 | 0.150 | 0.600 |
| MMT00006981 | Porcn | 0.150 | 0.609 |
| MMT00067776 | 0610010O12Rik | 0.161 | 0.620 |
| MMT00020602 | Spon2 | 0.108 | 0.621 |
| MMT00022932 | BC025446 | 0.179 | 0.635 |
| MMT00081543 | Serpina3m | 0.129 | 0.635 |
| MMT00001154 | 2600001J17Rik | 0.150 | 0.637 |
| MMT00035101 | Atp6v1a1 | 0.145 | 0.637 |
| MMT00070429 | Bace2 | 0.171 | 0.639 |
| MMT00053710 | Papss1 | 0.144 | 0.639 |
| MMT00043004 | Asf1b | 0.159 | 0.644 |
| MMT00075972 | Cyp2c55 | 0.147 | 0.649 |
| MMT00000887 | Gne | 0.140 | 0.654 |
| MMT00076051 | 2310014L17Rik | 0.208 | 0.654 |
| MMT00045344 | BC025600 | 0.156 | 0.656 |
| MMT00059782 | Ctsk | 0.145 | 0.657 |
| MMT00032264 | Nek2 | 0.126 | 0.658 |
| MMT00070618 | Npl | 0.161 | 0.659 |
| MMT00016157 | Gck | 0.166 | 0.660 |
| MMT00066819 | AW146242 | 0.147 | 0.668 |
| MMT00035158 | 5730469M10Rik | 0.154 | 0.682 |
| MMT00014927 | H2afz | 0.127 | 0.685 |
| MMT00048462 | Golph2 | 0.152 | 0.689 |
| MMT00036060 | Clcn5 | 0.164 | 0.698 |
| MMT00051292 | Egfr | 0.146 | 0.699 |
| MMT00056256 | Ephx1 | 0.131 | 0.708 |
| MMT00028949 | 1700012B18Rik | 0.185 | 0.709 |
| MMT00038075 | Arhgap21 | 0.143 | 0.715 |
| MMT00061203 | 9330129D05Rik | 0.147 | 0.718 |
| MMT00011089 | Cyp2c54 | 0.130 | 0.723 |
| MMT00077244 | mKIAA1236 | 0.151 | 0.724 |
| MMT00013585 | Actg | 0.153 | 0.726 |
| MMT00011446 | Ahcy | 0.135 | 0.729 |
| MMT00040982 | D19Wsu12e | 0.130 | 0.730 |
| MMT00082677 | Gbe1 | 0.144 | 0.731 |
| MMT00059202 | Rcn | 0.159 | 0.731 |
| MMT00074527 | Tk1 | 0.149 | 0.733 |
| MMT00016835 | Gpld1 | 0.098 | 0.735 |
| MMT00042966 | Lin7a | 0.173 | 0.735 |
| MMT00031650 | Eml1 | 0.126 | 0.740 |
| MMT00032545 | Tm4sf6 | 0.136 | 0.747 |
| MMT00063382 | Mcm2 | 0.121 | 0.752 |
| MMT00045329 | Nab2 | 0.150 | 0.755 |
| MMT00033624 | Dact2 | 0.133 | 0.759 |
| MMT00079874 | Tjp2 | 0.159 | 0.760 |
| MMT00019204 | Gys2 | 0.171 | 0.762 |
| MMT00037385 | Dnaja1 | 0.154 | 0.764 |
| MMT00017157 | Chc1l | 0.176 | 0.766 |
| MMT00013100 | 2810489O06Rik | 0.131 | 0.769 |
| MMT00003970 | Ctps | 0.133 | 0.772 |
| MMT00008578 | AA408451 | 0.171 | 0.773 |
| MMT00037403 | Tjp3 | 0.144 | 0.778 |
| MMT00081967 | Psmb10 | 0.129 | 0.817 |
| MMT00061273 | Osbpl3 | 0.143 | 0.819 |
| MMT00011257 | Fhit | 0.174 | 0.824 |
| MMT00030448 | 4833409A17Rik | 0.165 | 0.830 |
| MMT00033085 | Mme | 0.137 | 0.841 |
| MMT00018925 | Hamp | 0.176 | 0.842 |
| MMT00002002 | Unc5b | 0.132 | 0.853 |
| MMT00004264 | Abcd3 | 0.124 | 0.874 |
| MMT00047786 | Banp | 0.180 | 0.883 |
| MMT00039764 | Timp3 | 0.125 | 0.890 |
| MMT00062045 | Ywhah | 0.130 | 0.906 |
| MMT00016621 | LOC13909 | 0.142 | 0.913 |
| MMT00042811 | Pigr | 0.151 | 0.918 |
| MMT00052337 | Rgs5 | 0.141 | 0.950 |
| MMT00031178 | Mug1 | 0.151 | 0.955 |
| MMT00053241 | Olig2 | 0.129 | 0.955 |
| MMT00041166 | Heph | 0.137 | 0.964 |
| MMT00072646 | Nup210 | 0.162 | 0.976 |
| MMT00080984 | Gnai1 | 0.140 | 0.979 |